당뇨병 위험 분류 예측 프로젝트(이진분류) 2 : 이상치 처리 및 피처 엔지니어링
1. 기본 교차 성능 검증
데이터 전처리나 피처 엔지니어링을 하지 않은 상태에서 교차 검증을 수행하는 주된 목적은 "기준 성능(baseline performance)"을 설정하는 것입니다.
이 기준 성능은 후속적으로 적용되는 다양한 데이터 전처리나 피처 엔지니어링 기법의 효과를 명확하게 측정하고 평가하는 기준점 역할을 합니다.
즉, 이를 통해 특정 전처리나 피처 엔지니어링이 모델 성능에 어떤 영향을 미치는지 알 수 있습니다.
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import cross_val_score, cross_validate, KFold, StratifiedKFold
from sklearn.metrics import accuracy_score, recall_score, f1_score, precision_score, roc_auc_score, confusion_matrix
features_org = train.columns[1:-1]
train_x = train[features_org]
train_y = train['Outcome']
kf = StratifiedKFold(n_splits=4, shuffle=True, random_state=42)
display("####### 기본 교차 검증 성능 #########")
RF_model = RandomForestClassifier(random_state = 42)
cv_result = cross_validate(RF_model, train_x, train_y, cv=kf, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result = pd.DataFrame(cv_result, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
display(df_cv_result)
display(df_cv_result.describe().loc['mean',:].to_frame().T)
※ Stratified K-Fold
Stratified K-Fold 교차 검증은 K-Fold 교차 검증의 변형입니다.
기본적인 K-Fold 교차 검증은 데이터를 K개의 부분(즉, "fold")으로 나눈 다음, 각 부분을 검증 세트로 사용하고 나머지 부분을 학습 세트로 사용하여 모델을 학습 및 검증합니다.
그러나 K-Fold 교차 검증은 클래스 불균형을 고려하지 않습니다. 이로 인해 특정 폴드에서는 소수 클래스의 샘플이 거의 없을 수 있어, 모델의 성능 평가가 부정확할 수 있습니다.
반면, Stratified K-Fold 교차 검증은 각 폴드에서의 클래스 분포가 전체 데이터셋의 클래스 분포와 유사하도록 데이터를 분할합니다.
즉, 각 폴드는 전체 클래스의 비율을 대체로 유지하게 됩니다. 이는 각 클래스를 공정하게 표현하도록 하여, 모델의 성능 평가를 보다 정확하게 만듭니다.
따라서 stage2에서 step2 "Target 변수 class별 비율"에서 살펴본 바와 같이, 우리가 다루고 있는 이진 분류 문제에서는 class 0(정상)와 class 1(당뇨병)의 비율이 약 65% - 35% 수준이면 클래스 불균형이 있다고 판단되므로, Stratified K-Fold 교차 검증을 사용하는 것이 좋습니다.
※ 결과 해석
4개의 fold에서의 평균 정확도는 약 73.62%로 나타났습니다.
평균 정밀도(precision)는 약 64.76%로 나타났습니다. 이는 모델이 양성(당뇨병 환자)으로 예측한 결과 중 실제로 양성인 비율을 의미합니다.
평균 재현율(recall)은 약 54.39%로 나타났습니다. 이는 실제 양성 중에서 모델이 양성으로 예측한 비율을 의미합니다.
평균 F1 점수는 약 58.85%로 나타났습니다. F1 점수는 정밀도와 재현율의 조화 평균으로, 두 지표의 균형을 나타냅니다.
이 결과를 통해 RandomForestClassifier 모델의 교차 검증 성능을 파악할 수 있습니다.
2. IQR 기반 이상치
2.1 IQR 기반 이상치(Outlier) 검출
# 이상치를 탐지하는 함수 정의
def detect_outliers(dataframe, column):
Q1 = dataframe[column].quantile(0.25)
Q3 = dataframe[column].quantile(0.75)
IQR = Q3 - Q1
# IQR 기반으로 이상치 범위를 정의
lower_bound = Q1 - 1.5 * IQR
upper_bound = Q3 + 1.5 * IQR
# 이상치의 인덱스를 반환
return dataframe[(dataframe[column] < lower_bound) | (dataframe[column] > upper_bound)].index
# 숫자형 데이터만 대상으로 이상치 탐지
numeric_columns = train.select_dtypes(include=[np.number]).columns.tolist()
outliers_dict = {}
for column in features_org:
outliers = detect_outliers(train, column)
if len(outliers) > 0:
outliers_dict[column] = outliers
display(f"num of outlier of {column} : {len(outliers)}")
display(outliers_dict)
2.2 IQR 기반의 이상치 (Outlier) 중위값 대체 및 교차 검증 변화 확인
train_dout_median = train.copy()
#이상치를 중앙값으로 대체
for column, outlier_indices in outliers_dict.items():
median_value = train_dout_median[column].median()
train_dout_median.loc[outlier_indices, column] = median_value
train_dout_median_x = train_dout_median[features_org]
train_dout_median_y = train_dout_median['Outcome']
RF_model = RandomForestClassifier(random_state = 42)
# 이상치 처리 후 RandomForest를 사용한 교차 검증 성능 확인
cv_result_dout_median = cross_validate(RF_model, train_dout_median_x, train_dout_median_y, cv=kf, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_dout_median = pd.DataFrame(cv_result_dout_median, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
display(df_cv_result_dout_median)
display(df_cv_result_dout_median.describe().loc['mean',:].to_frame().T)
※ 결과 해석
모델의 교차 검증 성능은 평균 정확도를 기준으로 약 74.08%로 측정되었습니다.
이는 이전에 우리가 얻은 기본 교차 검증 성능인 73.62%보다 약간 더 높습니다.
이번에 IRQ 방식을 이용해 이상치를 처리하였는데, 이 방법을 사용하니 모델의 성능이 소폭 상승하였습니다.
하지만, 이 상승 폭이 그리 크지 않다는 점은 주목할 필요가 있습니다.
이것은 IRQ 기반의 이상치 처리 방법이 이 데이터셋에서는 모델의 성능을 크게 향상시키지 못했다는 것을 의미합니다.
2.3 IQR 기반의 이상치 (Outlier) 제거 및 교차 검증 성능 변화 확인
train_delete_outlier = train.copy()
# 원본 train 데이터에서 이상치 제거
rows_to_drop = set()
for column in features_org:
outliers_indices = detect_outliers(train_delete_outlier, column)
rows_to_drop.update(outliers_indices)
train_delete_outlier = train_delete_outlier.drop(rows_to_drop)
train_delete_outlier_x = train_delete_outlier[features_org]
train_delete_outlier_y = train_delete_outlier['Outcome']
# 이상치를 제거한 후 RandomForest를 사용한 교차 검증 성능 확인
cv_result_delete_outlier = cross_validate(RF_model, train_delete_outlier_x, train_delete_outlier_y, cv=kf, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_delete_outlier = pd.DataFrame(cv_result_delete_outlier, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
display(f"IQR 기반의 이상치 제거한 갯수 : {len(train) - len(train_delete_outlier)} 개, 비율 : {(len(train) - len(train_delete_outlier))/len(train) * 100.0}")
display(df_cv_result_delete_outlier)
display(df_cv_result_delete_outlier.describe().loc['mean',:].to_frame().T)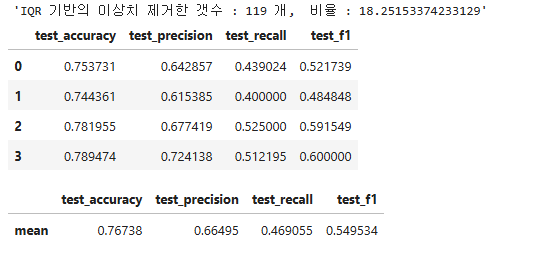
※ 결과 해석
모델의 교차 검증 성능은 평균 정확도를 기준으로 약 76.74%로 측정되었습니다.
이는 중위수 기반의 이상치 처리 방법(74.08%)과 비교했을 때, 그리고 기본 교차 검증 성능(73.62%)과 비교했을 때 모두 눈에 띄게 향상된 수치입니다.
그러나 이 방법을 사용하면서 데이터의 약 18.25%, 즉 119개의 데이터를 제거하게 되었습니다.
이는 전체 데이터의 상당량을 제거하는 것을 의미하며, 이러한 방식은 주의가 필요합니다.
데이터를 지나치게 많이 제거하게 되면, 우리가 가지고 있는 유용한 정보를 잃게 되며 이는 모델의 성능에 오히려 부정적인 영향을 미칠 수 있습니다.
특히, 실제 프로젝트에서는 테스트 데이터에도 유사한 이상치가 존재할 가능성이 높습니다.
학습 데이터에서 많은 양의 이상치를 제거하는 것은 실제 테스트 환경에서의 모델 성능을 왜곡시킬 수 있습니다.
이는 학습 데이터에서만 잘 작동하고 테스트 데이터에서는 성능이 떨어지는 모델을 만드는 결과를 가져올 수 있기 때문에 바람직하지 않은 접근 방법입니다.
3. Z-score 기반 이상치 제거 및 교차 검증 성능 변화 확인
※ Z-score(제트 스코어)
Z-score는 "표준 점수(Standard Score)"라고도 부릅니다.
이는 개별 데이터 포인트가 데이터셋의 평균에 비해 얼마나 멀리 떨어져 있는지를 나타내는 값입니다.
특히, Z-score는 데이터 포인트가 평균에서 표준 편차의 몇 배만큼 떨어져 있는지를 알려줍니다.
- Z=0: 데이터 포인트 X가 평균과 동일합니다.
- Z>0: 데이터 포인트 X가 평균보다 높습니다.
- Z<0: 데이터 포인트 X가 평균보다 낮습니다.
Z-score의 절대값이 3보다 큰 데이터 포인트는 보통 이상치로 간주됩니다.
from scipy import stats
# Z-score 기반 이상치 제거
z_scores = np.abs(stats.zscore(train_x))
threshold = 3 # 이 값을 조절하여 이상치로 간주되는 임계점을 설정합니다.
train_zscore = train.copy()[(z_scores < threshold).all(axis=1)]
display(f"z_score 기반의 이상치 제거한 갯수 : {len(train) - len(train_zscore)} 개, 비율 : {(len(train) - len(train_zscore))/len(train) * 100.0}")
# 데이터 업데이트
train_zscore_x = train_zscore[features_org]
train_zscore_y = train_zscore['Outcome']
display("####### Z-score 기반 이상치 제거 후 교차 검증 성능 #########")
RF_model = RandomForestClassifier(random_state=42)
cv_result_zscore = cross_validate(RF_model, train_zscore_x, train_zscore_y, cv=kf, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_zscore = pd.DataFrame(cv_result_zscore, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
display(df_cv_result_zscore)
display(df_cv_result_zscore.describe().loc['mean',:].to_frame().T)
※ 결과 해석
우리의 모델의 교차 검증 성능은 평균 정확도를 기준으로 약 76.85%로 측정되었습니다.
이는 IQR 기반의 이상치 제거 방법을 사용했을 때보다 정확도가 높아졌음을 의미합니다.
더욱이, 제거된 데이터의 수도 69개로 크게 줄었습니다.
이 결과를 통해, 평균에서 표준 편차의 3배 이상 떨어져 있는 데이터가 모델의 성능에 부정적인 영향을 미쳤다는 것을 알 수 있습니다.
또한, Z-score를 활용한 이상치 제거 방법이 이 데이터셋에서 효과적임을 확인할 수 있습니다.
그러나 이를 통해, 이상치 제거 방법이 데이터셋마다, 문제마다 다르게 적용되어야 하며, 여러 방법을 실험해보는 것이 중요하다는 알 수 있습니다.
이상치를 처리하면 모델의 성능을 향상시키는 데 도움이 될 수 있지만, 동시에 중요한 정보를 잃을 가능성도 있으므로 신중하게 접근해야 합니다.
4. SMOTE 통한 데이터 불균형
※ KMeansSMOTE
KMeansSMOTE는 SMOTE(Synthetic Minority Over-sampling Technique)를 기반으로 하는 오버샘플링 기법으로, KMeans 알고리즘을 사용하여 소수 클래스 데이터를 클러스터링하고, 각 클러스터에서 새로운 데이터를 생성합니다.
데이터 누출을 방지하기 위해, 오버샘플링을 교차 검증의 각 반복에서 학습 데이터에만 적용하는 점이 중요합니다.
이렇게 하면, 검증 데이터가 오버샘플링 과정에서 사용된 정보를 포함하지 않게 되므로, 과적합을 방지하고 더 신뢰할 수 있는 성능 평가를 수행할 수 있습니다.
- KMeansSMOTE를 사용하여 smote_kmeans 객체를 생성합니다.
- kf.split을 사용하여 학습 데이터와 검증 데이터의 인덱스를 분할합니다.
- iloc 메소드를 사용하여 학습 데이터와 검증 데이터를 분할합니다.
- smote_kmeans.fit_resample 메소드를 사용하여 학습 데이터에만 SMOTE 오버샘플링을 적용합니다.
- 오버샘플링된 학습 데이터로 RandomForest 모델을 학습합니다. fit 메소드를 통하여 학습을 수행합니다.
- 검증 데이터에 대한 예측을 수행합니다. predict 메소드를 통하여 예측을 수행합니다.
- accuracy_score를 사용하여 성능 지표를 계산하고, 이를 accuracies 리스트에 추가합니다.
- np.mean을 사용하여 성능 지표의 평균을 계산합니다.
from imblearn.over_sampling import SMOTE, BorderlineSMOTE,KMeansSMOTE,SVMSMOTE
from sklearn.metrics import accuracy_score
rf_model_resampled_ = RandomForestClassifier(random_state = 42)
# 교차 검증을 수동으로 구현하기 위한 리스트 초기화
accuracies = []
kf = StratifiedKFold(n_splits=4, shuffle=True, random_state=42)
smote_kmeans = KMeansSMOTE(random_state=42)
for train_idx, val_idx in kf.split(train_zscore_x, train_zscore_y):
# 학습 데이터와 검증 데이터 분할
X_train, X_val = train_zscore_x.iloc[train_idx], train_zscore_x.iloc[val_idx]
y_train, y_val = train_zscore_y.iloc[train_idx], train_zscore_y.iloc[val_idx]
# 학습 데이터에만 SMOTE 오버샘플링 적용
X_train_resampled, y_train_resampled = smote_kmeans.fit_resample(X_train, y_train)
# 오버샘플링된 학습 데이터로 모델 학습
rf_model_resampled_.fit (X_train_resampled, y_train_resampled)
# 검증 데이터에 대한 예측
val_predictions = rf_model_resampled_.predict(X_val)
# 성능 지표 계산
accuracies.append(accuracy_score(val_predictions, y_val))
# 성능 지표의 평균 계산
mean_accuracy = np.mean(accuracies)
display(f"accuracy of each Fold : {accuracies}")
display(f"accuracy of mean_accuracy : {mean_accuracy}")
※ 결과 해석
각 교차 검증에서의 정확도는 각각 약 76.33%로서, Z-score를 통한 이상치 제거 후의 교차 검증에서의 평균 정확도(76.85%)보다 감소하였습니다.
이를 통해, 이 데이터셋에는 KMeansSMOTE를 활용한 오버샘플링이 모델의 성능 향상을 이끌어내지는 못했음을 알 수 있습니다.
이는 불균형 데이터에 대한 오버샘플링이 항상 성능 향상을 보장하지 않으며, 특정 데이터셋에는 오히려 성능 저하를 가져올 수 있음을 보여줍니다.
5. 피처 엔지니어링
5.1 feature pair의 조합 연산 통한 새로운 feature 생성
- itertools의 combinations 메소드를 사용하여 주어진 특성 목록 features_subset에서 크기가 2인 모든 조합을 생성합니다.
- 새로운 특성 'feature1_feature2_Ratio'를 생성하는 과정에서 나눗셈 연산을 수행하므로, 분모가 0이 되지 않도록 주의해야 합니다.
replace 메소드를 사용하여 0을 중앙값으로 대체합니다. 여기서 중앙값은 median 메소드를 사용하여 계산합니다. - cross_val_score 함수를 사용하여 주어진 모델과 특성을 사용하여 교차 검증을 수행합니다.
pd.concat을 사용하여 기존 특성과 새로운 특성을 결합하고, mean 메소드를 사용하여 평균 점수를 계산합니다.
import itertools
# 기존 피처의 조합을 생성하고, 새로운 피처를 추가한 후 각 조합에 대한 교차 검증 점수를 계산합니다.
# 그 중에서 가장 높은 정확도를 가진 피처만 선택합니다.
# RandomForest 모델과 KFold 객체를 생성합니다.
rf_model = RandomForestClassifier(random_state = 42)
# 주어진 피처의 조합을 생성합니다.
features_subset = ['Insulin', 'Age', 'BMI']
feature_combinations = list(itertools.combinations (features_subset, 2))
cv_scores = {}
for feature_pair in feature_combinations:
feature1, feature2 = feature_pair
train_try = train_zscore_x.copy()
# 새로운 피처를 추가합니다.
train_try[f'{feature1}_{feature2}_Diff'] = train_try[feature1] - train_try[feature2]
train_try[f'{feature1}_{feature2}_Sum'] = train_try[feature1] + train_try[feature2]
train_try[f'{feature1}_{feature2}_Ratio'] = train_try[feature1] / (train_try[feature2].replace(0, train_try[feature2].median()))
features_to_evaluate = [f'{feature1}_{feature2}_Diff', f'{feature1}_{feature2}_Sum', f'{feature1}_{feature2}_Ratio']
# 각 피처에 대한 교차 검증 점수를 계산합니다.
feature_scores = {}
for feature in features_to_evaluate:
scores = cross_val_score(rf_model, pd.concat([train_zscore_x, train_try[feature]], axis=1), train_zscore_y, cv=kf, scoring='accuracy')
feature_scores[feature] = scores.mean()
# 가장 높은 점수를 가진 피처만 선택합니다.
best_feature = max(feature_scores, key=feature_scores.get)
cv_scores[best_feature] = feature_scores[best_feature]
display(f"cv_scores[{best_feature}] : {cv_scores[best_feature]}")
# 점수를 기준으로 정렬합니다.
# 먼저, cv_scores 딕셔너리의 각 항목을 리스트로 변환합니다.
items = list(cv_scores.items())
# 그 다음, 이 리스트를 정렬합니다. 각 항목은 (key, value) 쌍이므로, item[1]을 사용하여 값을 기준으로 정렬합니다.
sorted_items = sorted(items, key=lambda item: item[1], reverse=True)
# 이제 이 정렬된 리스트를 다시 딕셔너리로 변환합니다.
cv_scores = dict(sorted_items)
display(cv_scores)
※ 결과 해석
Insulin, Age 조합에서는 'Insulin_Age_Sum'이라는 새로운 피처를 추가하였을 때 모델의 정확도가 약 78.4%로, 이상치 제거 후 기본 교차 검증의 정확도인 76.85%보다 약 1.55%포인트 성능 향상이 있었습니다.
이는 두 피처의 합이라는 새로운 정보가 모델의 타겟 클래스를 예측하는 데 도움이 되었음을 의미합니다.
Insulin, BMI 조합에서는 'Insulin_BMI_Ratio'라는 새로운 피처를 추가하였을 때 모델의 정확도가 약 78.2%로, 이상치 제거 후 기본 교차 검증의 정확도인 76.85%보다 약 1.35%포인트 성능 향상이 있었습니다.
이는 인슐린과 BMI의 비율이라는 새로운 정보가 모델이 타겟 클래스를 예측하는 데 도움이 되었음을 의미합니다.
Age, BMI 조합에서는 'Age_BMI_Ratio'라는 새로운 피처를 추가하였을 때 모델의 정확도가 약 76.7%로, 이상치 제거 후 기본 교차 검증의 정확도인 76.85%보다 약 0.15%포인트 성능 저하가 있었습니다.
이는 나이와 BMI의 비율이라는 새로운 정보가 모델이 타겟 클래스를 예측하는 데 도움이 되지 않았음을 의미합니다.
이런 결과는 새로운 피처를 생성할 때, 모든 피처의 조합이나 연산이 유의하거나 효과적이지 않을 수 있음을 보여줍니다.
5.2 feature pair의 조합 연산 통한 feature 추가하여 교차 검증 성능 확인
유용하다고 판단한 두개의 피쳐 'Insulin_Age_Sum', 'Insulin_BMI_Ratio'를 train_zscore에 추가하고, 교차 검증 성능을 확인
- 'Insulin'과 'Age'의 합을 나타내는 새로운 특성을 생성합니다.
- 그리고 'BMI' 값이 0인 경우 중앙값으로 대체하여 나눗셈에서 무한대 값이 발생하는 것을 방지합니다. 이후 'Insulin'과 'BMI'의 비율을 나타내는 특성을 생성합니다.
'BMI' 값이 0인 경우 중앙값으로 대체하는 것은 나눗셈에서 분모가 0이 되는 상황을 피하기 위한 조치입니다. - 기존 데이터 프레임에 새로 생성된 특성을 추가합니다.
from sklearn.ensemble import RandomForestClassifier
# 피쳐 생성
train_try = train_zscore.copy()
train_try['Insulin_Age_Sum'] = train_try['Insulin'] + train_try['Age']
train_try['BMI'] = train_try['BMI'].replace(0, train_try['BMI'].median())
train_try['Insulin_BMI_Ratio'] = train_try['Insulin'] / train_try['BMI']
# 피쳐 추가
train_prep = train_zscore.copy()
train_prep['Insulin_Age_Sum'] = train_try['Insulin_Age_Sum']
train_prep['Insulin_BMI_Ratio'] = train_try['Insulin_BMI_Ratio']
train_prep_y= train_prep['Outcome']
train_prep_x = train_prep.drop(['ID', 'Outcome'], axis=1)
RF_model_prep = RandomForestClassifier(random_state=42)
cv_result_prep = cross_validate(RF_model_prep, train_prep_x, train_zscore_y, cv=kf, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_prep = pd.DataFrame(cv_result_prep, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
display(df_cv_result_prep)
display(df_cv_result_prep.describe().loc['mean',:].to_frame().T)
display(train_prep_x.head(5))

5.3 test 데이터에 동일한 피쳐 생성, 추가 하기
test['Insulin_Age_Sum'] = test['Insulin'] + test['Age']
test['BMI'] = test['BMI'].replace(0, test['BMI'].median())
test['Insulin_BMI_Ratio'] = test['Insulin'] / test['BMI']
test.head(5)