당뇨병 위험 분류 예측 프로젝트(이진분류) 4 : 비결측치 그룹에 대한 데이터 전처리 및 피처 엔지니어링
1. 인슐린 결측치가 없는 정상 train 데이터(train_normal) 추출
from sklearn.preprocessing import StandardScaler
from sklearn.ensemble import RandomForestClassifier
# feature set 정의
features_org = list(train.columns)[1:-1]
# train 데이터에서 Insulin이 0이 아닌 데이터 추출
train_normal = train.copy()
train_normal = train_normal.loc[train_normal['Insulin'] != 0]
train_normal_x = train_normal[features_org]
train_normal_y = train_normal['Outcome']
display(train_normal.head(5))
2. Z-score 기반의 이상치 제거
from scipy import stats
features_org = train.columns[1:-1]
train_normal_x = train_normal[features_org]
# Z-score 기반 이상치 제거
z_scores = np.abs(stats.zscore(train_normal_x))
threshold = 3 # 이 값을 조절하여 이상치로 간주되는 임계점을 설정합니다.
train_zscore = train_normal.copy()[(z_scores < threshold).all(axis=1)]
display(f"z_score 기반의 이상치 제거한 갯수 : {len(train_normal) - len(train_zscore)} 개, 비율 : {(len(train_normal) - len(train_zscore))/len(train_normal) * 100.0}")'z_score 기반의 이상치 제거한 갯수 : 26 개, 비율 : 7.784431137724551'3. train_normal에 대한 기본 교차 검증 성능 확인
RandomForestClassifier를 사용하여 인슐린 값이 결측치가 없는 데이터셋(train_normal)에 대하여, RandomForestClassifier 모델로 기본 교차 검증 성능을 확인해보겠습니다.
from sklearn.model_selection import cross_val_score, cross_validate, KFold,StratifiedKFold
kf = StratifiedKFold(n_splits=4, shuffle=True, random_state=42)
train_normal_x = train_normal[features_org]
train_normal_y = train_normal['Outcome']
RF_model_normal = RandomForestClassifier(random_state = 42)
cv_result_normal = cross_validate(RF_model_normal, train_normal_x, train_normal_y, cv=kf, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_normal = pd.DataFrame(cv_result_normal, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
display(df_cv_result_normal)
display(df_cv_result_normal.describe().loc['mean',:].to_frame().T)
4. 상관관계 통해 새로운 변수 생성에 대한 아이디어 얻기
점 이연 상관계수와 p-value를 계산하며, 그 결과를 시각화한 후에, 새로운 변수를 어떻게 생성해야 할지 고민해보겠습니다.
import seaborn as sns
import matplotlib.pyplot as plt
from scipy.stats import pointbiserialr
correlation_org_lst, correlation_dealout_lst = [], []
p_value_org_lst, p_value_dealout_lst = [], []
# 점 이연 상관계수 계산 및 출력
for feature in features_org:
correlation_org, p_value_org = pointbiserialr(train_normal[feature], train_normal['Outcome'])
correlation_org_lst.append(correlation_org)
p_value_org_lst.append(p_value_org)
# 데이터프레임 생성
correlation_dict = {'Feature': features_org,
'correlation_org': correlation_org_lst,
'p_value_org' : p_value_org_lst }
correlation_df = pd.DataFrame(correlation_dict)
display(correlation_df)
plt.figure(figsize=(18, 6))
plt.subplot(1,3,1)
sns.barplot(x='Feature', y='correlation_org', data=correlation_df)
plt.gca().set_title("Point Biserial Correlation [original train]")
plt.gca().set_xticklabels(features_org, rotation=30)
plt.subplot(1,3,2)
sns.barplot(x='Feature', y='p_value_org', data=correlation_df)
plt.gca().set_xticklabels(features_org, rotation=30)
plt.gca().set_title("p_value [original train]")
plt.subplot(1,3,3)
sns.heatmap(train_normal[features_org].corr(), cmap = "coolwarm", annot= True )
plt.tight_layout()
plt.show()

※ 결과 해석
데이터에서 각 피처와 결과 (Outcome)의 상관 관계를 분석한 결과, BloodPressure와 DiabetesPedigreeFunction만 p-value가 상대적으로 높은값을 가지므로 다른 피처와 비교했을 때 상관 관계 유의성이 떨어집니다.
BloodPressure나 DiabetesPedigreeFunction 등의 feature와 다른 피처와의 조합을 고려하여 새로운 피처를 생성하는 아이디어를 생각해 볼 수 있습니다.
예를 들면, 혈압과 다른 건강 지표와의 비율이나 조합 등을 고려하여 새로운 의미 있는 피처를 생성해 볼 수 있습니다.
상관 관계 유의성이 상대적으로 떨어지는 BloodPressure 피처를 중심으로 피처 엔지니어링을 진행하면 더 좋은 모델 성능을 얻을 수 있을 것입니다.
5. train_normal 데이터에 feature 조합 통한 새로운 feature 생성 (1)
'BloodPressure', 'BMI' 조합으로 피처 연산 방법을 이용하여 새로운 feature 생성
- 'BloodPressure'와 'BMI'를 더하거나 빼서 새로운 피처를 생성
- 'BloodPressure'를 'BMI'로 나눈 값을 새로운 피처로 생성, 이때 'BMI'가 0인 값은 중위수(median)값으로 대체합니다.
from sklearn.ensemble import RandomForestClassifier
# 피처 후보 생성
train_normal_try1 = train_normal.copy()
train_normal_try1['BloodPressure_BMI_Diff'] = train_normal_try1['BloodPressure'] - train_normal_try1['BMI']
train_normal_try1['BloodPressure_BMI_Sum'] = train_normal_try1['BloodPressure'] + train_normal_try1['BMI']
train_normal_try1['BMI'] = train_normal_try1['BMI'].replace(0, train_normal_try1['BMI'].median())
train_normal_try1['BloodPressure_BMI_Ratio'] = train_normal_try1['BloodPressure'] / train_normal_try1['BMI']
train_normal_try1_x = train_normal_try1.drop('Outcome', axis=1)
features_to_evaluate = ['BloodPressure_BMI_Diff', 'BloodPressure_BMI_Sum', 'BloodPressure_BMI_Ratio']
rf_model = RandomForestClassifier(random_state = 42)
# 교차 검증 성능 비교
cv_scores = {}
for feature in features_to_evaluate:
train_normal_add_x = train_normal[features_org].copy()
train_normal_add_x[feature] = train_normal_try1[feature]
scores = cross_val_score(rf_model, train_normal_add_x, train_normal_y, cv=kf, scoring='accuracy')
cv_scores[feature] = scores.mean()
display(f"accuracy : {cv_scores}")"accuracy : {'BloodPressure_BMI_Diff': 0.769399024670109, 'BloodPressure_BMI_Sum': 0.8023164084911073, 'BloodPressure_BMI_Ratio': 0.7903399311531842}"
※ 결과 해석
생성된 세 개의 피처가 기존의 피처에 추가될 경우 교차 검증 정확도는
- 'BloodPressure_BMI_Diff': 76.94%
- 'BloodPressure_BMI_Sum': 80.23%
- 'BloodPressure_BMI_Ratio': 79.03% 입니다.
'BloodPressure'와 'BMI'를 더한 값이 baseline 교차 검증 성능(79.04%)보다 높습니다.
이는 "혈압"과 "체질량지수"가 '동시에' 높을 때 당뇨 발병에 영향이 큰 것으로 해석할 수 있습니다.
따라서 인슐린 결측치가 없는 정상 데이터에서는 'BloodPressure_BMI_Sum'을 새로운 피처로 생성합니다.
6. train_normal 데이터에 feature 그룹화 통한 새로운 feature 생성하기
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import cross_val_score, cross_validate, KFold
from sklearn.preprocessing import LabelEncoder
train_normal_try2 = train_normal.copy()
# BloodPressure의 사분위수 계산
q1 = np.percentile(train_normal_try2['BloodPressure'], 25)
q2 = np.percentile(train_normal_try2['BloodPressure'], 50)
q3 = np.percentile(train_normal_try2['BloodPressure'], 75)
q4 = np.percentile(train_normal_try2['BloodPressure'], 100)
# BloodPressure를 2개의 범주로 나누기
q_BloodPressure_lst = [ 0, q1, q4]
BloodPressure_labels_train = pd.cut(train_normal_try2['BloodPressure'], bins = q_BloodPressure_lst, labels = ['q1', 'q2'])
# LabelEncoder로 범주형 데이터 인코딩
le_train_ = LabelEncoder()
train_normal_try2['BloodPressure_cat'] = le_train_.fit_transform(BloodPressure_labels_train)
# "BloodPressure_cat" 범주에 따른 "DiabetesPedigreeFunction"의 빈도수 계산
train_normal_try2['DiabetesPedigreeFunction_by_BloodPressure_cat'] = train_normal_try2.groupby('BloodPressure_cat')['DiabetesPedigreeFunction'].transform('count')
# BloodPressure_cat" 범주에 따른 "DiabetesPedigreeFunction"의 빈도수 dict형태로 backup
DPF_counts_by_BloodPressure_cat_train = train_normal_try2.groupby('BloodPressure_cat')['DiabetesPedigreeFunction'].count().to_dict()
display(DPF_counts_by_BloodPressure_cat_train)
# 피처가 추가된 데이터로 교차 검증 수행
train_normal_try2_x = train_normal_try2.drop(['ID', 'Outcome','BloodPressure_cat'], axis=1)
rf_model = RandomForestClassifier(random_state = 42)
# 결과 출력
cv_result_try = cross_val_score(rf_model, train_normal_try2_x, train_normal_y, cv=kf, scoring='accuracy')
display(f"accuracy : { cv_result_try.mean()}")
display(train_normal_try2.head(5))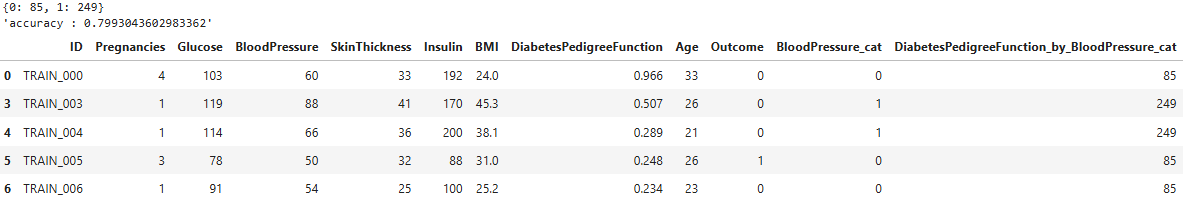
※ 결과 해석
새로 추가된 피처를 포함한 데이터로 RandomForest 분류기를 사용하여 교차 검증을 수행한 결과, 정확도는 약 79.93%입니다.
baseline 교차 검증 성능(0.790)보다 향상된 결과로서, 타겟 변수인 당뇨병 발병 여부에 대한 설명력 높은 의미 있는 피처일 가능성이 매우 높습니다.
7. 새로 생성한 feature 추가하여 교차 검증 성능 확인 (2)
이번에는 'BloodPressure'와 'DiabetesPedigreeFunction' 두 특성을 조합하여 타겟 변수와의 의미 있는 연관성을 찾아보겠습니다.
'BloodPressure' 특성은 이전 분석에서 타겟과의 상관관계가 매우 낮았으므로, 이를 개선하기 위해 새로운 피처 생성 방법을 탐색합니다.
이 과정을 통해 기존에 상관관계가 낮았던 두 특성을 조합하여 타겟 변수와의 연관성을 높이는 새로운 피처를 생성하고, 모델의 예측 성능을 향상시키는 시도해보겠습니다.
train_normal_prep = train_normal.copy()
train_normal_prep['BloodPressure_BMI_Sum'] = train_normal_try1['BloodPressure_BMI_Sum']
train_normal_prep['DiabetesPedigreeFunction_by_BloodPressure_cat'] = train_normal_try2['DiabetesPedigreeFunction_by_BloodPressure_cat']
# 점이연 상관 관계 유의성 없는 'BloodPressure', 'DiabetesPedigreeFunction' 제거
train_normal_prep = train_normal_prep.drop('BloodPressure', axis=1)
train_normal_prep = train_normal_prep.drop('DiabetesPedigreeFunction', axis=1)
train_normal_prep_x = train_normal_prep.drop(['ID','Outcome'], axis=1)
# RandomForestClassifier로 오버샘플링된 데이터에 대한 교차 검증
RF_model_prep = RandomForestClassifier(random_state=42)
cv_result_normal_prep = cross_validate(RF_model_prep, train_normal_prep_x, train_normal_y, cv=kf, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_normal_prep = pd.DataFrame(cv_result_normal_prep, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
display(df_cv_result_normal_prep)
display(df_cv_result_normal_prep.describe().loc['mean',:].to_frame().T)
display(train_normal_prep_x.head(5))
※ 결과 해석
교차 검증 결과로 얻은 통계를 보면, 평균 정확도는 약 79.92%입니다.
이는 추가된 피처들이 모델의 성능에 긍정적인 영향을 미치고 있음을 시사합니다.
8. test_normal 데이터에 새로운 feature 생성하기
test_normal = test.copy()
test_normal = test_normal.loc[test_normal['Insulin'] != 0]
# 'BloodPressure_BMI_Sum' 생성
test_normal_prep = test_normal.copy()
test_normal_prep.loc[:,'BloodPressure_BMI_Sum'] = test_normal_prep['BloodPressure'] + test_normal_prep['BMI']
# 'DiabetesPedigreeFunction_by_BloodPressure_cat' 생성
BloodPressure_labels_test = pd.cut(test_normal_prep['BloodPressure'], bins = q_BloodPressure_lst, labels = ['q1', 'q2'])
test_normal_prep['BloodPressure_cat'] = le_train_.fit_transform(BloodPressure_labels_test)
# "BloodPressure_cat" 범주 (train 데이터, train_normal_try2 기준)에 따른 "DiabetesPedigreeFunction"의 빈도수 (train 데이터, train_normal_try2의 통계치) 로 대입
test_normal_prep['DiabetesPedigreeFunction_by_BloodPressure_cat'] = test_normal_prep['BloodPressure_cat'].apply(lambda x: DPF_counts_by_BloodPressure_cat_train.get(x))
# 불필요한 feature 제거
test_normal_prep = test_normal_prep.drop('BloodPressure_cat', axis=1)
test_normal_prep = test_normal_prep.drop('BloodPressure', axis=1)
test_normal_prep = test_normal_prep.drop('DiabetesPedigreeFunction', axis=1)
display(test_normal_prep.head(7))