1. 전처리 및 피쳐 엔지니어링 완료된 데이터 읽어오기 1
'Insulin'이 결측치가 없는 정상 데이터인 'train_normal'에 대해 전처리와 피처 엔지니어링을 적용하여 저장한 'train_normal_prep.csv'와 'test_normal_prep.csv' 파일을 불러오겠습니다.
이 파일들을 각각 독립변수와 종속변수로 분할합니다
import pandas as pd
from sklearn.preprocessing import StandardScaler
train_normal_prep = pd.read_csv('train_normal_prep.csv')
train_normal_prep_y = train_normal_prep['Outcome']
train_normal_prep_x = train_normal_prep.drop(['ID', 'Outcome'], axis=1)
test_normal_prep = pd.read_csv('test_normal_prep.csv')
test_normal_prep_x = test_normal_prep.drop('ID', axis=1)
features_normal_selected = train_normal_prep_x.columns
display(train_normal_prep.head(5))
display(test_normal_prep.head(5))
2. 전처리 및 피쳐 엔지니어링 완료된 데이터 읽어오기 2
'Insulin'이 결측치인 데이터에 대해서도 같은 작업을 수행하겠습니다.
전처리와 피처 엔지니어링이 적용된 'train_abnormal_prep.csv'와 'test_abnormal_prep.csv' 파일을 로드하겠습니다.
이 파일들을 각각 독립변수와 종속변수로 분할합니다.
train_abnormal_prep = pd.read_csv('train_abnormal_prep.csv')
train_abnormal_prep_y = train_abnormal_prep['Outcome']
train_abnormal_prep_x = train_abnormal_prep.drop(['ID', 'Outcome'], axis=1)
features_abnormal_selected = train_abnormal_prep_x.columns
######################################################################################################################
test_abnormal_prep = pd.read_csv('test_abnormal_prep.csv')
test_abnormal_prep_x = test_abnormal_prep.drop('ID', axis=1)
display(train_abnormal_prep.head(5))
display(test_abnormal_prep.head(5))
3. 교차 검증 성능 재확인
'train_normal_prep'에 대해서는 StratifiedKFold를, 'train_abnormal_prep'에 대해서는 KFold를 사용하여 교차 검증을 진행하겠습니다.
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import cross_val_score, cross_validate, KFold,StratifiedKFold
kf_normal = StratifiedKFold(n_splits=4, shuffle=True, random_state=42)
kf_abnormal = KFold(n_splits=4, shuffle=True, random_state=42)
display("############ [ RandomForestClassifier ] ######################################")
print("\n")
display("인슐린 정상 데이터 (test_normal_prep)에 대한 교차 검증 성능")
RF_model_normal = RandomForestClassifier(random_state = 42)
cv_result_normal = cross_validate(RF_model_normal, train_normal_prep_x, train_normal_prep_y, cv=kf_normal, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_normal = pd.DataFrame(cv_result_normal, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
display(df_cv_result_normal.describe().loc['mean',:].to_frame().T)
display("인슐린 결측 데이터 (test_abnormal_prep)에 대한 교차 검증 성능")
RF_model_abnormal = RandomForestClassifier(random_state = 42)
cv_result_abnormal = cross_validate(RF_model_abnormal, train_abnormal_prep_x, train_abnormal_prep_y, cv=kf_abnormal, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_abnormal = pd.DataFrame(cv_result_abnormal, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
display(df_cv_result_abnormal.describe().loc['mean',:].to_frame().T)
4. 인슐린 정상 데이터에 RFECV 에 의한 feature selection 적용하기
※ Recursive Feature Elimination with Cross-Validation (RFECV)
RFECV는 모델의 성능을 기반으로 가장 중요한 특성을 선택하는 방법입니다.
RFECV는 기본적으로 모델의 중요도를 기반으로 가장 중요하지 않은 특성부터 순차적으로 제거해 나가며, 모델의 성능을 교차 검증으로 평가합니다.
이런 과정을 통해 모델의 성능을 최대로 끌어낼 수 있는 특성의 개수를 결정하게 됩니다.
또한, 특성 선택 후에는 이 선택된 특성을 기반으로 모델의 하이퍼파라미터를 튜닝하는 과정이 이어지게 됩니다.
특성 선택을 통해 불필요한 특성을 제거하면 모델이 중요한 특성에 집중하게 되고, 과적합을 방지하면서도 좋은 성능을 얻을 수 있습니다.
이후 하이퍼파라미터 튜닝을 통해 모델의 성능을 더욱 최적화할 수 있습니다.
- RFECV를 이용한 특성 선택: RFECV 클래스를 사용하여 특성 선택을 진행합니다. RandomForestClassifier를 추정기로 사용하고, step=1, cv=kf_normal, scoring='accuracy'로 설정합니다.
- RFECV에 데이터 적용: fit 메소드를 사용하여 RFECV에 데이터를 적용하고 특성을 선택합니다
- 선택된 특성에 대한 데이터 준비: RFECV의 support_ 속성을 사용하여 선택된 특성에 대한 데이터를 준비합니다
from sklearn.feature_selection import RFECV
import time
start_time = time.time()
# RFECV를 이용한 특성 선택
rf_model_normal = RandomForestClassifier(random_state=42)
rfecv_normal = RFECV(estimator=rf_model_normal, step=1, cv=kf_normal, scoring='accuracy')
rfecv_normal = rfecv_normal.fit(train_normal_prep_x, train_normal_prep_y)
# 선택된 특성에 대한 데이터 준비
train_normal_prep_x_selected = train_normal_prep_x[train_normal_prep_x.columns[rfecv_normal.support_]]
# 교차 검증
cv_result_normal_selected = cross_validate(rf_model_normal, train_normal_prep_x_selected, train_normal_prep_y, cv=kf_normal, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_normal_selected = pd.DataFrame(cv_result_normal_selected, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
# 선택된 특성 출력 및 교차 검증 결과 출력
features_normal_selected = train_normal_prep_x.columns[rfecv_normal.support_]
display(f"selected features of train_normal_prep : {features_normal_selected}")
display(df_cv_result_normal_selected)
display(df_cv_result_normal_selected.describe().loc['mean',:].to_frame().T)
end_time = time.time()
display(f"total time elapse : {end_time-start_time}")

5. 인슐린 정상 데이터에 대한 하이퍼 파라미터 튜닝
import time
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.model_selection import GridSearchCV
start_time = time.time()
# 파라미터의 분포 설정
param_dist = {
'n_estimators': np.arange(100, 300, 50),
'max_depth': [ 8, 16 ]
}
# feature selection
rf_model_normal = RandomForestClassifier(random_state = 42)
# GridSearchCV 설정
search_rf_normal = GridSearchCV(estimator=rf_model_normal, param_grid =param_dist,
cv=kf_normal, verbose=2)
search_rf_normal.fit(train_normal_prep_x[features_normal_selected], train_normal_prep_y)
# 최적의 파라미터와 그 때의 점수 출력
print(f"Best parameters: {search_rf_normal.best_params_}")
print(f"Best cross-validated score: {search_rf_normal.best_score_}")
# 결과를 데이터프레임으로 변환
results_normal = pd.DataFrame(search_rf_normal.cv_results_)
# n_estimators와 max_depth에 따른 피벗 테이블 생성
pivot_table_normal = results_normal.pivot_table(values='mean_test_score',
index='param_n_estimators',
columns='param_max_depth')
end_time = time.time()
display(f"total time elapse : {end_time-start_time}")
6. 인슐린 정상데이터에 대한 그리드 서치(GridSearchCV) 결과 시각화
# 히트맵으로 시각화
plt.figure(figsize=(12, 4))
plt.subplot(1,2,1)
plt.title("Mean Test Score vs. n_estimators and max_depth")
sns.heatmap(pivot_table_normal, annot=True, cmap='YlGnBu', fmt='.4g')
plt.subplot(1,2,2)
for col in pivot_table_normal.columns:
plt.plot(pivot_table_normal[col], label=f'max_depth = {col}') # 각 max_depth 값에 대한 선 그리기
plt.title("Score vs. n_estimators for different max_depth values")
plt.legend(title="max_depth") # 범례의 제목을 'max_depth'로 설정
plt.xlabel("n_estimators")
plt.ylabel("Mean Test Score")
plt.tight_layout()
plt.show()
7. 인슐린 결측치 데이터에 RFECV 에 의한 feature selection 적용
start_time = time.time()
# RFECV를 이용한 특성 선택
rf_model_abnormal = RandomForestClassifier(random_state=42)
rfecv_abnormal = RFECV(estimator=rf_model_abnormal, step=1, cv=kf_abnormal, scoring='accuracy')
rfecv_abnormal = rfecv_normal.fit(train_abnormal_prep_x, train_abnormal_prep_y)
# 선택된 특성에 대한 데이터 준비
train_abnormal_prep_x_selected = train_abnormal_prep_x[train_abnormal_prep_x.columns[rfecv_abnormal.support_]]
# 교차 검증
cv_result_abnormal_selected = cross_validate(rf_model_normal, train_abnormal_prep_x_selected, train_abnormal_prep_y, cv=kf_abnormal, scoring=['accuracy', 'precision', 'recall', 'f1'])
df_cv_result_abnormal_selected = pd.DataFrame(cv_result_abnormal_selected, columns=['test_accuracy', 'test_precision', 'test_recall', 'test_f1'])
# 선택된 특성 출력 및 교차 검증 결과 출력
features_abnormal_selected = train_abnormal_prep_x.columns[rfecv_abnormal.support_]
display(f"selected features of train_abnormal_prep : {features_abnormal_selected}")
display(df_cv_result_abnormal_selected)
display(df_cv_result_abnormal_selected.describe().loc['mean',:].to_frame().T)
end_time = time.time()
display(f"total time elapse : {end_time-start_time}")
8. 인슐린 결측 데이터에 대한 하이퍼파라미터 튜닝
start_time = time.time()
kf = KFold(n_splits=4, shuffle=True, random_state=42)
# 파라미터의 분포 설정
param_dist = {
'n_estimators': np.arange(100, 300, 50),
'max_depth': [8, 16 ]
}
rf_model_abnormal = RandomForestClassifier(random_state=42)
search_rf_abnormal = GridSearchCV(estimator=rf_model_abnormal, param_grid =param_dist,
cv=kf_abnormal, verbose=2)
search_rf_abnormal.fit(train_abnormal_prep_x[features_abnormal_selected], train_abnormal_prep_y)
# 최적의 파라미터와 그 때의 점수 출력
print(f"Best parameters: {search_rf_abnormal.best_params_}")
print(f"Best cross-validated score: {search_rf_abnormal.best_score_}")
# 결과를 데이터프레임으로 변환
results_abnormal = pd.DataFrame(search_rf_abnormal.cv_results_)
# n_estimators와 max_depth에 따른 피벗 테이블 생성
pivot_table_abnormal = results_abnormal.pivot_table(values='mean_test_score',
index='param_n_estimators',
columns='param_max_depth')
9. 인슐린 결측 데이터에 대한 그리드 서치(GridSearchCV) 결과 시각화
# 히트맵으로 시각화
plt.figure(figsize=(12, 4))
plt.subplot(1,2,1)
plt.title("Mean Test Score vs. n_estimators and max_depth")
sns.heatmap(pivot_table_abnormal, annot=True, cmap='YlGnBu', fmt='.4g')
plt.subplot(1,2,2)
for col in pivot_table_abnormal.columns:
plt.plot(pivot_table_abnormal[col], label=f'max_depth = {col}') # 각 max_depth 값에 대한 선 그리기
plt.title("Score vs. n_estimators for different max_depth values")
plt.legend(title="max_depth") # 범례의 제목을 'max_depth'로 설정
plt.xlabel("n_estimators")
plt.ylabel("Mean Test Score")
plt.tight_layout()
plt.show()
end_time = time.time()
display(f"total time elapse : {end_time-start_time}")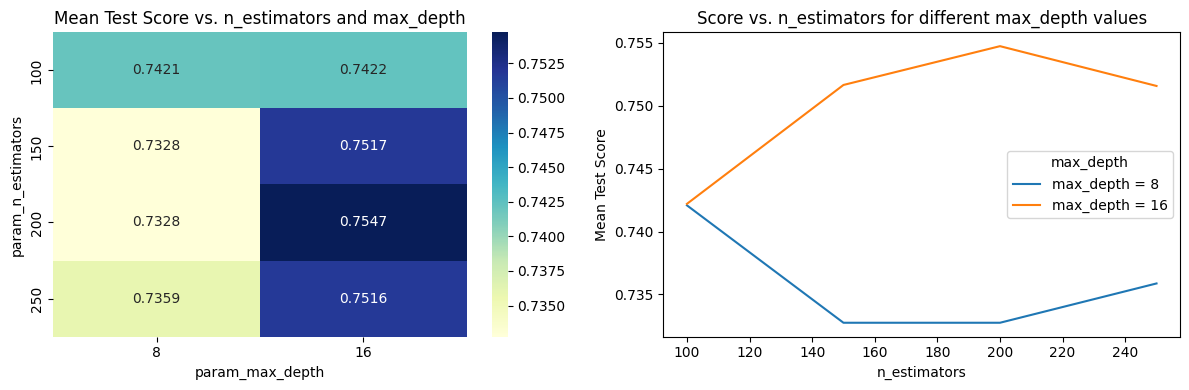
'total time elapse : 31.969544172286987'10. best model로 예측 및 예측 결과 합쳐서 저장
from sklearn.metrics import accuracy_score
search_rf_normal.best_estimator_.fit(train_normal_prep_x[features_normal_selected], train_normal_prep_y)
pred_normal_arr = search_rf_normal.best_estimator_.predict(test_normal_prep[features_normal_selected])
search_rf_abnormal.best_estimator_.fit(train_abnormal_prep_x[features_abnormal_selected], train_abnormal_prep_y)
pred_abnormal_arr = search_rf_abnormal.best_estimator_.predict(test_abnormal_prep[features_abnormal_selected])
ID_normal_arr = test_normal_prep['ID'].values
pred_normal = pd.DataFrame({'ID': ID_normal_arr, 'Outcome' : pred_normal_arr})
ID_abnormal_arr = test_abnormal_prep['ID'].values
pred_abnormal = pd.DataFrame({'ID': ID_abnormal_arr, 'Outcome' : pred_abnormal_arr})
# 두 DataFrame 합치기
submission = pd.concat([pred_normal, pred_abnormal])
submission = submission.sort_values(by ='ID').reset_index(drop=True)
submission.to_csv('submission.csv', index=False)'머신러닝 > 머신러닝: 실전 프로젝트 학습' 카테고리의 다른 글
| 인구 소득 예측 프로젝트 2 : 결측값 처리 (1) | 2024.12.31 |
|---|---|
| 인구 소득 예측 프로젝트 1 : 데이터 분석 (1) | 2024.12.30 |
| 당뇨병 위험 분류 예측 프로젝트(이진분류) 5 : 결측치 그룹에 대한 데이터 전처리 및 피처 엔지니어링 (0) | 2024.12.29 |
| 당뇨병 위험 분류 예측 프로젝트(이진분류) 4 : 비결측치 그룹에 대한 데이터 전처리 및 피처 엔지니어링 (0) | 2024.12.29 |
| 당뇨병 위험 분류 예측 프로젝트(이진분류) 3 : 결측치 데이터 분석 (1) | 2024.12.29 |